データセットの仕様を刷新しました
Starrydataのデータセットのスキーマを刷新したので共有します。
過去のスキーマ(Version 1.0、以下v1)のデータセットはGithub、新しいスキーマ(Version 2.0、以下v2)はFigshareで公開している023年7月25日以降のデータセットになります。
変更の動機
v1では全データを1つのCSVファイルで提供していましたが、2023年にはそのファイル容量が3GBを超え、メモリ容量が少ないPCではファイルを開くのが困難になるようになりました。具体的なこれまでの全データファイルの容量の推移は下記になります。
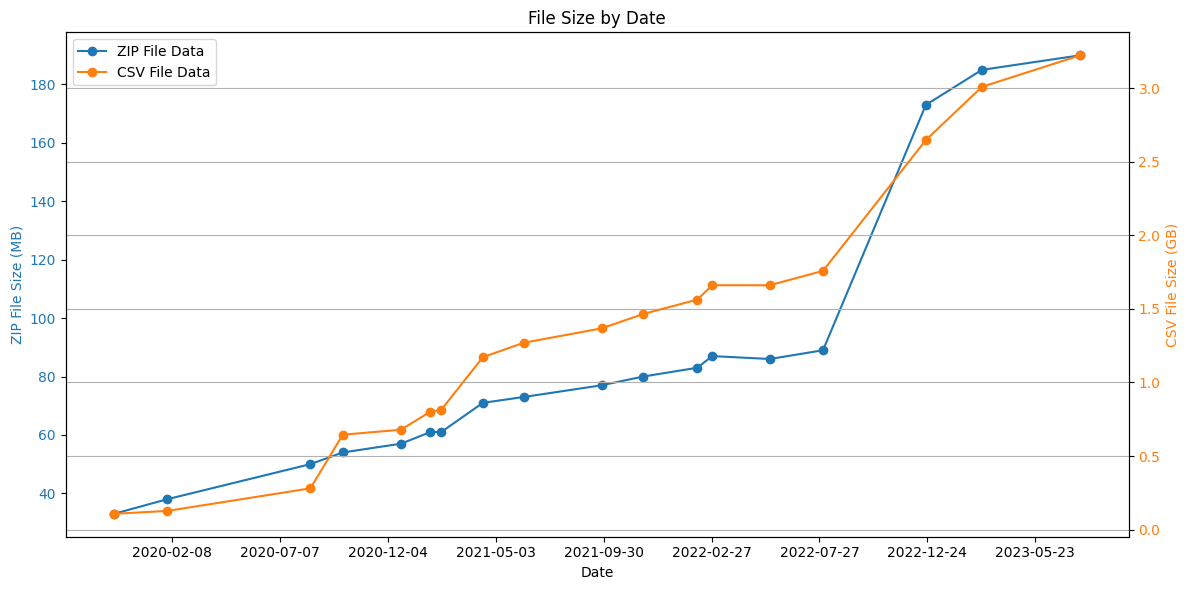
このファイル容量を減らすことがデータセットのスキーマ変更の動機になります。容量削減のいくつかの対策に加えて、品質向上のためにもいくつか対策をしているので、下記に紹介していきます。
削減対策1:1ファイルから3ファイルに分割(正規化)
v1ではXYグラフのカーブの値から書誌情報、サンプルデータまで全てを1ファイルにしていました。しかしその結果、冗長な箇所が増えてしまいデータ容量の増加につながっていました。
そのため、いわゆる正規化をすることで冗長性を無くすことでファイル容量を小さくしました。具体的には、カーブデータ、論文データ、サンプルデータの3つに分け、それぞれ「all_curves.csv」「all_papers.json」「all_samples.csv」というファイル名にしました。削減対策の結果は以下の通りです。
| 対策:1ファイルから3ファイルに分割(正規化) |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
1.3GB |
109MB |
約92% |
|
メモリ使用量 |
971MB |
169MB |
約83% |
想定通り寄与が大きく、ファイル容量を90%以上も削減することができました。
削減対策2:XY値は1行1データから1行1カーブへ
下記のようなXYグラフがあったとします。
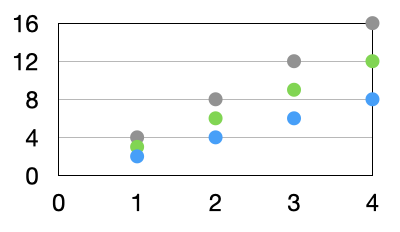
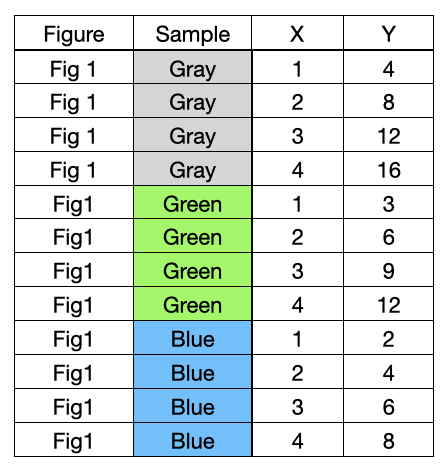
v1ではこれらの12のデータ点を1点1行としていました。下記のような形式です。
これをv2ではカーブで1行としています。下記のような形式です。
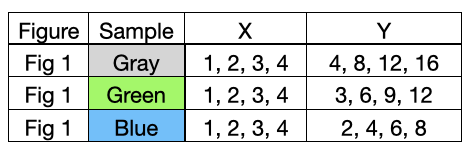
メリットとして、カーブ単位で補間をしたい場合などに、解析者が点をグループ化してカーブにする必要がなくなりました。v1の時はFigure IDとSample IDを使っていました。また、行数が減ることで他の列の冗長な箇所が減りファイル容量削減にも寄与しています。その結果は下記の通りです。
| 対策:XY値は1行1データから1行1カーブへ |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
444MB |
109MB |
約76% |
|
メモリ使用量 |
1,792MB |
169MB |
約91% |
こちらも想定通り大きく削減率に貢献してくれています。
削減対策3:物理量は列ではなく行として収録
v1では温度などの物理量は列として収録していました。具体的には下記のような形になります。Property A, B, C・・・と物理量の数だけ列が存在していました。
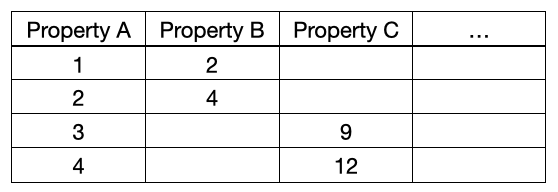
それをv2では下記のように物理量名は物理量Xと物理量Yの列に入れるようにしました。StarrydataのデータセットがXYグラフを対象に集めていたためこのような変更が可能でした。
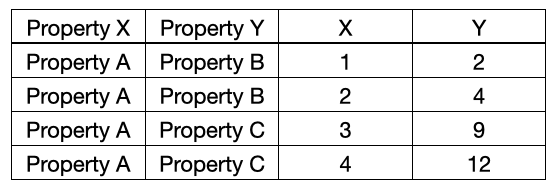
削減率の結果は下記の通りです。
| 対策:物理量は列ではなく行として収録 |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
111MB |
109MB |
約2% |
|
メモリ使用量 |
257MB |
169MB |
約34% |
区切り文字がカンマ形式のCSVファイルにおいて空白セルが大量にあったとしても、空白セルの数だけカンマは増えますが、カンマ自体は1byteのみの増加だったため、想定通りCSVファイル容量の削減率は2%と大きく寄与しませんでした。一方でメモリ使用量は約34%と小さくない寄与率があったため、こちらも有効な削減対策になったと考えています。
削減対策4:有効数字を7桁に設定
Starrydataに登録されてあるデータは論文中のグラフからの抽出データであり、グラフの画像の解像度に限界がある以上、取得できる有効数字にも限界があると考えています。なぜならデータ点を抽出時に使うのはモニターであり、その画面を構成する点の最小単位である「ピクセル」以下の点の抽出は難しいからです。厳密には画像の縮尺を変更することで、さらに数倍程度までは精度を高めることができるとは思いますが。
桁数が多いとデータ容量の増加にも繋がってしまう一方で、不要な桁数は削除することでデータセットを軽くすることができるため、有効数字を設定することにしました。
その結果は下記の通りです。
| 対策:有効数字を7桁に設定 |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
120MB |
109MB |
約9% |
|
メモリ使用量 |
190MB |
169MB |
約11% |
ファイル容量とメモリ使用量、共に10%前後と大きな寄与率ではないですが、小さくもないので一定の寄与がありました。
ちなみに有効数字を7桁とした理由ですが、データ収集する際に用いる画面は、データ収集者によりますが、大きくても4K(3,840x2,160)と仮定して、さらに画像を3倍拡大して抽出した場合、1万ピクセルでの抽出が可能です。仮にX軸がリニアスケールであり、その値が1から10,000だった場合、正確に有効数字5桁の抽出は可能でしょう。そのため有効数字は少なくとも5桁あれば十分かと考えていますが、グラフによっては、X軸の値が1.010001から1.020000という場合も存在します。現在は、このような状況も考慮して余裕を持って有効数字7桁としていますが、収集する対象プロジェクトや対象グラフによっては、この設定でも問題が出る可能性があるので、今後も状況に応じて変更する可能性はあります。
削減対策5:収録する物理量を22個に絞る
※ 2023/10/12追記
2023/10/12以降のデータセットでは物理量の絞り込みを廃止し、全ての物理量を表示するように変更しました。理由は、メインの3つのプロジェクト以外の物理量もデータ解析したいという声があり、今回の下の結果にも示している通り、対策前後のデータ量の差異は12%であり、データ量削減の寄与が他の対策と比べて大きくないため、利便性を考慮した際に廃止した方が良いと考えたためです。
Starrydataはユーザー参加型のシステムであり、一般ユーザーでも自由にマスターとなる物理量を追加することができます。そのため、運営チーム以外のユーザーにもデータを入れていただくことが出来ました。しかし収集ルールを運営チームがきちんと定義できていなかったため、重複する物理量が発生してしまいました。そのため、今回のデータセットでは運営チームがデータを再度確認して、一定の品質を担保できる物理量のみ出力するため、物理量は絞っています。具体的な物理量はデータセットのREADMEファイルをご覧ください。
こちらの対策は品質向上が主目的になりますが、もちろん削減対策にもなっていますので結果を下記に示します。
| 対策:収録する物理量を絞る |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
124MB |
109MB |
約12% |
|
メモリ使用量 |
193MB |
169MB |
約12% |
ファイル容量とメモリ容量共に12%でした。こちらの結果からStarrydataのデータは運営チームで収集しているものが多くの割合を占めていることが分かります。
まとめ
|
ファイル容量削減率 |
メモリ使用量削減率 |
|
|
1ファイルから3ファイルに分割(正規化) |
約92% |
約83% |
|
XY値は1行1データから1行1カーブへ |
約76% |
約91% |
|
物理量は列ではなく行として収録 |
約2% |
約34% |
|
有効数字を7桁に設定 |
約9% |
約11% |
|
物理量を絞る収録する物理量を絞る |
約12% |
約12% |
|
合計 |
約97%(3.3GB -> 111MB) |
約98%(10.55GB -> 176MB) |
3.3GBあったCSVファイルを111MBまで削減することができ、メモリ使用量も約98%削減することにできたので、以前の仕様に比べて、スペックの低いPCでも解析がスムーズにできるようになったと思います。今後もより多くの方に使いやすくするための開発を進めていきたいです。
補足
今回の検証は下記環境にて行いました。
- macOS Monterey 12.2.1
- Python 3.10.12
- pandas 2.0.2
利用したStarrydataのデータは2023-08-30 00:00:01 JST+0900時点のものです。
追記 2023.09.14
「削減対策3:物理量は列ではなく行として収録」ですが、削減対策5の収録する物理量を絞った上で行っており、正確にv1との比較ができていないことに気づいたので、追加で確認しました。
結果はこちらです。
| 対策:物理量 (266個) は列ではなく行として収録 |
対策なし |
対策あり |
削減率 |
|
CSVファイル容量 |
256MB |
119MB |
約54% |
|
メモリ使用量 |
1,633MB |
162MB |
約90% |
v1では物理量は全て採用しており、2023.8.30時点で266種類ありました。Starrydataでは収集対象XYデータになるため、そのうち値が入ってるのは2つなので、99%が空白セルになりますが、CSVファイルは「,」区切りフォーマットを採用しており、「,」が少なくとも1行あたり266個あります。そのため、物理量を30個に絞った状態で測定した時にCSVファイル容量が約2%しか削減できませんでしたが、今回は約54%削減できています。さらにメモリ使用量の削減率も約34%から約90%に上げることができました。
追記 2023.09.20
今回、データセットの仕様を変更するにあたり、データセット生成のスクリプトも大幅に改善しました。v1ではPythonのpymongoライブラリを利用して一度全てのデータベースの内容をメモリに展開してデータベースのコレクションの結合を行なっていましたが、v2ではCSVに整形する処理を除いた全ての処理をMongoDBのAggregation Pipelineを利用しました。またMongoDBのインデックスも見直しました。そのため、不要なデータ読み込みとデータ操作を減らすことができ、メモリ使用量を20GB超から2GB弱へ、実行時間を1時間超から3分へと短縮することができました。
その結果、データセットの生成はこれまでローカルのスペックの高いマシン(メモリ16GB以上)でしか行っていたかったデータセット生成処理を費用を抑えたバッチサーバー(1コア・メモリ2GB)に移行することができ、日時でのデータセット生成を実現することができました。
この記事はNIMSエンジニアの間藤 智也が執筆しました。




